한참전에 윈도우 기준으로 로컬 PC에 BLAST를 설치하고 돌리는 글을 이전 블로그에 쓴 적이 있다. 그런데 그것도 벌써 한 9년 전의 일이고, BLAST 자체도 기본적으로 바뀌지는 않았지만 BLAST+ 라는 형태로 바뀌었다. 그래서 업데이트하는 느낌으로 OS X 에서 Local BLAST를 설치하고 돌리는 과정을 적어보고자 한다. (윈도우는 어떻게 해염 하려면 옛날 글 을 참조하셈)
1. 다운로드
요기에 가서 dmg 포맷의 설치파일을 받는다. 세상 많이 좋아졌다. dmg 포맷의 설치파일이 나오니..
제일 아랫넘이다.
2. 설치
패키지 선택하고
오오 GUI Installer! (그러면 뭐해 어차피 나중엔 다 커맨드 라인인데)
여튼 이렇게 하면 쨘~ 하고 설치를 끝낸다. 그런데 대체 어디에 설치를 해놓은 거냐.
3. 설정
이렇게 설치를 하면 기본적으로 /usr/local/ncbi/blast 내에 파일을 복사해 놓는다. 
옛날에 커맨드 라인 BLAST (지금 현재 Legacy BLAST라고 이야기하는) 를 설치해 본 사람이라면 명령어가 좀 틀려졌다는 느낌을 받을 것이다. 즉 이전에는 blastall 이라는 커맨드에서 blastp, blastn 등을 지정했지만 이제는 별도의 명령어가 등장했다.
여튼 여기서 추가로 설정을 해주어야 한다. blast 실행파일이 있는 /usr/local/ncbi/blast/bin에 대한 PATH는 설치과정에서 되니 (/etc/path.d 에 해놓는다) 할 필요가 없지만, blast가 db 파일을 찾을 디렉토리와 환경변수 ($BLASTDB)는 따로 만들어 두는 것이 좋다.
/usr/local/ncbi/blast에서
su mkdir db
로 db 디렉토리를 만들고, ~/.profile 을 편집한다.
vim ~/.profile
난 vi 못씀! 하면 다른 걸 써라. TextEdit이든 Sublime Text이건..
로 .profile에 들어가서
BLASTDB=/usr/local/ncbi/blast/db
export BLASTDB
를 추가.
source ~/.profile
을 하거나 쉘을 재시동.
그 다음에 db를 설치한다. 만들어진 db를 아까 만든 db 디렉토리에 넣어도 되지만 FASTA 형식으로 된 시퀀스를 가지고 db를 설치할 수도 있다. 이걸 해보자.
cd $BLASTDB
pwd
/usr/local/ncbi/blast/db
만들어놓은 DB를 다운받는것도 좋지만 걍 FASTA 형식의 시퀀스를 받아서 db를 만들어보자.
sudo wget ftp://ftp.ncbi.nlm.nih.gov/blast/db/FASTA/pdbaa.gz
해서 다운받고
sudo gunzip pdbaa.gz
압축풀고, 이제 blast용 db를 만들어보자. 이전에는 formatdb라는 것을 썼지만 요즘은 makeblastdb 라는 것을 써야한다고 한다.
makeblastdb -help
USAGE
makeblastdb [-h] [-help] [-in input_file] [-input_type type]
-dbtype molecule_type [-title database_title] [-parse_seqids]
[-hash_index] [-mask_data mask_data_files] [-mask_id mask_algo_ids]
[-mask_desc mask_algo_descriptions] [-gi_mask]
[-gi_mask_name gi_based_mask_names] [-out database_name]
[-max_file_sz number_of_bytes] [-logfile File_Name] [-taxid TaxID]
[-taxid_map TaxIDMapFile] [-version]
….
긴데, 알아야 될 옵션은 두가지뿐.
-in 에서는 blastdb를 만들 입력파일 (여기서는 pdbaa)
-dbtype 에서는 시퀀스의 종류 (핵산의 경우에는 ‘nucl’, 단백질의 경우에는 ‘prot’)
sudo makeblastdb -in pdbaa -dbtype prot
명령을 내리면..
Building a new DB, current time: 02/22/2015 01:26:10
New DB name: pdbaa
New DB title: pdbaa
Sequence type: Protein
Deleted existing BLAST database with identical name.
Keep Linkouts: T
Keep MBits: T
Maximum file size: 1000000000B
Adding sequences from FASTA; added 75499 sequences in 3.41043 seconds.
pdbaa.pin, pdbaa.psq 라는 파일이 생기면 완료.
4. BLAST
이제 BLAST search를 날려봅시다. 검색할 대상이 있는 시퀀스가 저장된 디렉토리로 가서, 가령 Arp2.fasta 식으로 뭐 이런 시퀀스가 텍스트로 저장되어 있는 디렉토리로 이동했다고 치고, 여기서
blastp -query Arp2.fasta -db pdbaa
와 같이 query 뒤에 search하려는 파일, -db 뒤에 검색하려는 db (위의 예에서는 아까 만든 db인 pdbaa) 를 부르면
BLASTP 2.2.30+
Reference: Stephen F. Altschul, Thomas L. Madden, Alejandro A.
Schaffer, Jinghui Zhang, Zheng Zhang, Webb Miller, and David J.
Lipman (1997), “Gapped BLAST and PSI-BLAST: a new generation of
protein database search programs”, Nucleic Acids Res. 25:3389-3402
….
식으로 화면에 출력.
저장하려면
blastp -query Arp2.fasta -db pdbaa -out outfile
or
blastp -query Arp2.fasta -db pdbaa > out
포맷을 바꾸어서 hit 의 목록만 표시하려면
blastp -query Arp2.fasta -db pdbaa -out outfile -outfmt 7
하면
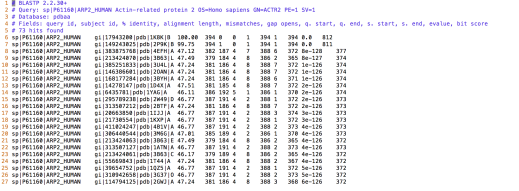
여튼, 구체적인 옵션은 blastp -help 하면 나옴. BLAST Parsing은 여기를 참조. 지금 21세기도 15년이 다 되었는데 BLAST Parser 따위는 새로 짜지마라. 제발…


